Índice
- O Curso R
-
- Tutoriais
-
- Apostila
-
- 6. Testes de Hipótese (em preparação!)
- Exercícios
-
- Material de Apoio
-
- Área dos Alunos
-
- Cursos Anteriores
-
IBUSP
Outras Insitutições
Linques
Visitantes
Outras Insitutições
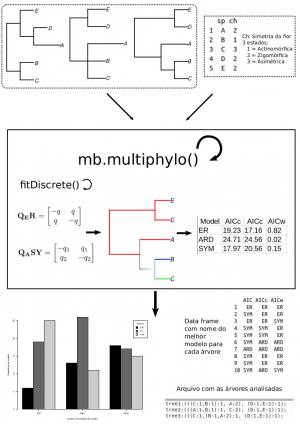
A reconstrução de estado ancestral é um método que permite entender a evolução de um caracter morfológico, neste caso discreto (e.g. Simetria da flor), quantificando as transformações entre os estados do caracter (e.g. actinomórfica, zigomórfica) sobre uma árvore filogenética. Existem duas aproximações para fazer reconstrução de estado ancestral, uma delas é estimando o estado mais provável para cada nó numa filogenia usando a função de Máxima Verossimilhança; a outra opção, amplamente usada atualmente, é uma aproximação Bayesiana que simula múltiplas reconstruções de estado ancestral sobre uma filogenia e estima a probabilidade posterior da cada um dos estados de caracter ocorrer em cada um dos nós da filogenia. O primeiro passo da análise é escolher o modelo de evolução que melhor explica a transformação entre os estados do carácter (q). Existem três modelos baseados no modelo mais básico para caracteres discretos que é o MK:
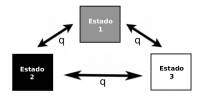
ER: As taxas de transformação entre estados são iguais, q12 = q21.
ARD: Todas as taxas de transformação entre caracteres são diferentes
SYM: Aplica para caracteres com mais de dois estados, donde as taxas de transformação são: q12 = q21, e q12 != q13.
Os métodos bayesianos permitem incorporar a incerteza da reconstrução; no entanto existem diferentes tipos de incerteza que podem ser tidos em conta. Fazer múltiplas simulações da reconstrução de estado ancestral numa filogenia contém a incerteza da reconstrução, mais é possível também fazer as simulações sobre uma amostra de árvores para incorporar a incerteza da reconstrução filogenética no análise de evolução dos caracteres. Phytools é um pacote desenvolvido por Liam Revel (2012) que contém múltiplas funções para fazer as análises ditas. No entanto o primeiro é testar o ajuste dos modelos de evolução morfológica numa amostra de árvores, e decidir qual deles usar para fazer as simulações.
A função permite ao usuário escolher uma amostra de árvores filogenéticas e avaliar o ajuste dos diferentes modelos de evolução de caracteres discretos para cada uma das árvores. Além disso irá retornar um sumario (numérico e gráfico) do ajuste, baseado no critério de preferência do usuário (e.g. AIC, AICc). A saída da função pode ser utilizada como entrada da função make.simmap de phytools para gerar as simulações de evolução do caracter sobre cada filogenía.
bm.multiphylo(trees, matrix, subsample = 0, stype = NULL, criterion = c(“AIC“, “AICc”, “AICw”), delta = 4)
trees = Objeto de clase multiphylo contendo múltiplas árvores filogenéticas
matrix = Data frame com codificação de caracteres morfológicos
subsample = Vetor com um valor numérico entre 2 e o número total de árvores que contém trees - 1
stype = Tipo de re-amostragem das árvores, pode ser escolhidas as n primeiras, n últimas ou n árvores ao acaso
criterion = Critério de seleção do modelo AIC, AICc ou AICw
delta = Diferença entre os valores do critério de seleção do melhor modelo com respeito ao segundo modelo. É um valor numérico entre 0 e infinito; no entanto existem valores reportados na literatura que são sugeridos 2 (Alencar et al., 2017), 4 (Harmon, 2018). Para o AICw será escolhido aquele modelo com maior valor
Pacotes requeridos: Para rodar a função é preciso instalar e carregar os pacotes ape, geiger, maps e phytools. Se não, a função não roda e envía a mensagem “O pacote X não está instalada no seu computador”.
trees = Objeto de clase multiphylo com árvores filogenéticas em notação parentética. Se não, a função não roda e envía a mensagem “Seu arquivo de árvores não posso ser lido, revise a notação das árvores além da extensão do arquivo”
matriz = O data frame debe conter uma coluna com o nome dos táxons e outra coluna com o caracter morfológico de interesse; caso que o data frame tenha mais de dois colunas, o usuário tem que espicificar qual delas quer analisar. Se não, a função não roda e envía a mensagem “Seu arquivo de caracteres não posso ser lido. No caso de ter mais de dois colunas revise se você definiu a coluna que contem o caracter que quer analisar”
1. Carregar os pacotes ape, geiger, maps e phytools
2. Ler o arquivo de arvores
3. Ler a matriz de dados morfológicos
4. Extrair a lista dos nomes dos táxons inclusos nos dos arquivos e comparar-las.
5. Usar um if caso que as listas de táxons não sejam iguais nem compatíveis, para a função e envía uma mensagem “Os táxons na matriz de dados e nas árvores filogenéticas não são compativeis”
5a. No Else, usar um novo if caso que as listas de táxons não sejam iguais porém sejam compatíveis faça o seguente:
drop.tip do pacote ape, dentro de um for com contador i desde 1 até número de árvores, para remover os táxons que não tem dados morfológicos no arquivo de árvores filogenéticas
6. Seja com os arquivos originais o com os novos objetos criados (árvores e matriz) use um For com contador i desde 1 até número de árvores, para aplicar a função fitDiscrete do pacote phytools para cada uma das árvores filogenéticas e com cada um dos modelos de evolução num contador j desde 1 até 3 que é o número de modelos para avaliar.
6a. Crie um vetor para guardar o valor de cada um dos critérios de avaliação dos modelos, para cada árvore analisada. Serão criados 9 vetores
6b. Crie um data frame com 10 colunas uma com o ID de cada árvore, e as 9 restantes são os vetores criados no ponto 6a.
7. Indexando pelas colunas do data frame para agrupas as colunas que contem informação de cada um dos critérios (e.g AIC), usar a função apply sobre as linhas para encontrar o seguente:
8. Sobre o novo data frame calcule a diferença (delta) entre os dois valores para cada um dos critérios (AIC e AICc)
9. Usar um if para fazer seleção do modelo baseado no valor de delta que o usuário escolheu. No entanto, ainda não sei muito bem como vou escolher o modelo mais simples caso que a diferença entre os modelos seja menor que a diferença escolhida. Tenho pensado criar uma matriz com os valores do critério escolhido para cada um dos modelos, e o delta nas colunas, e a primeira linha com o número de parâmetros de cada modelo, assim caso que delta seja menor ao número escolhido por o usuário a função pega o modelo com menor número de parâmetros, que é o modelo mais simples
10. Crie duas novas colunas no data frame do ponto 6b com a informação do modelo escolhido usando os critérios AIC e AICc
11. Sobre o data frame do punto 6b cheio, faça um barplot com a frequência relativa de cada um dos modelos ser o melhor modelo usando os diferentes critérios de seleção
12. Usar um if baseado no valor que o usuário escolheu para o argumento criterion, e indexar o data frame cheio sobre as colunas de interesse
13. Por fim, crie uma lista com o data frame do ponto 12 e com o objeto multiphylo criado no ponto 5a que contém as árvores filogenéticas usadas pela análise.
14. RETORNA a lista criada no ponto 13
A função tem como saída um gráfico de barras (barplot) com a frequência relativa de árvores para os quais cada um dos modelos de evolução de caracter foram escolhidos como o melhor modelo. Um arquivo .tre com a amostra de árvores analisada (caso que o usuário deseje fazer o análise com uma sub-amostra). Finalmente a função retorna um data frame com número de linhas igual ao número de árvores analisadas, e 5 colunas, uma com o ID de cada árvore, 3 contendo os valores para cada um dos critérios de seleção dos modelos de evolução (ARD, ER e SYM) e a última coluna com a nome do melhor modelo estimado para cada árvore, baseado em cada um dos critérios
Alencar LRV, Martins M, Burin G & Quental TB. 2017. Arboreality constrains morphological evolution but not species diversification in vipers. Proceedings of the Royal Society B: Biological Sciences 284: 20171775.
Harmon LJ. 2018. Phylogenetic comparative methods: Learning from trees. Available online: https://lukejharmon.github.io/pcm/
############################################
PCA (Principal Component Analysis) é uma análise multivariada exploratória que permite reduzir um conjunto amplo de variaveis a umas poucas que consigam explicar a maior parte da variação contida nos dados, i.e. é uma tecnica de redução das dimensões num conjunto de dados. As variaveis escolhidas podem ser analisadas depois usando ferramentas de estatística univariada visando responder perguntas sobre sua relação, diferençãs na variaveis dadas por um fator, entre outras.
A função usa o PCA para fazer uma seleção de variaveis potencialmente interessantes, baseada na quantidade de variação explicada (“eigenvector”) por cada uma das variaveis de um conjunto de dados. Além disso a função calcula a correlação entre as variaveis candidatas para evitar redundância na hora de explicar a variação dos dados.
var.selec (data, vrange = 0.7, cor = TRUE)
data = data frame contendo mais de 3 variaveis de tipo numérico
vrange = vetor numérico de 0 até 1. Representa a porcentagem de variação explicada por cada uma das variaveis
cor = vetor lógico que determina qual matriz de distança vai ser usada na análise de PCA. Se existem direfenças na magnitude dos valores para as variaveis analisadas, recomenda-se usar cor = TRUE, para que a matriz de correlação seja usada. Caso contrario cor = FALSE, a matriz usada é de covariação.
data = Os valores faltantes NAs serão removidos antes da análise
1. Ler o arquivo de dados
2. Usar um if para testar se o data frame contém dados faltantes; caso que tenha, enumera-los e remove-los, e envía uma mensagen “Sua matriz de dados contém X NAs que serão removidos da análise”
3. Fazer um PCA, e guardar os resultados num objeto de clase lista
4. Indexar o objeto do ponto 3 para pegar os valores dos “eigenvector” de cada uma das variaveis para os dois componentes que explicam a maior parte da variação dos dados
5. Criar um objeto com as variaveis que explicam uma porcentagem >= vrange (dado pelo usuário)
6. Usar um for para testar a correlação entre as variaveis escolhidas, e guardar o coeficiente de correlação, além do valor p num data frame
7. Retorna uma lista contendo três elementos: um data frame com os eigenvectors das variaveis escolhidas e duas matrices com resultados dos testes de correlação
Retorna uma lista contendo os seguentes elementos: um data frame com os nomes das variaveis escolhidas e seus eigenvectors correspondentes, uma matriz com os valores dos coeficientes de correlação para cada par de variaveis, uma matriz com os valores de p para o test de correlação entre cada par de variaveis. Além de retornar o gráfico do PCA
Olá Sandra. Nitidamente você dedicou bastante energia em construir a sua proposta A e como resultado eu acho que ela está boa e fazível. Não consigo avaliar se os objetos que as outras funções que você mencionou retornam ou pedem estão compatíveis com como você descreveu a sua função porque não conheço as ferramentas. Entretanto, o nível de detalhamento que você deu me convenceu que você sabe do que você está falando e, portanto, eu confio a você a responsabilidade sobre essa questão. Acho que sua proposta é ambiciosa, mas dado o seu perfil de aluna penso que você consegue sem grandes problemas.
A sua proposta B não parece ter recebido tanta atenção, mas isso é natural. Acho que você já está bem adiantada com a proposta A e, portanto, deveria investir nela. Mas entenda que eu estou passando para você parte da responsabilidade sobre o conteúdo da função e os que tipo de objetos que essa função pede de entrada e de saída.
Sobre a sua dúvida no ponto 9. da sua proposta A, acho que a o que você está pensando pode dar certo sim. Se você precisar de ajuda com qualquer coisa da função, pode me procurar na sala 243 da ecologia ou manda um email
— Bruno Travassos 2019/06/14 11:46
Olá Bruno,
Muito obrigada pelo retorno. A verdade não sei se vou conseguir desenvolver essa proposta sem grandes problemas, mas vou tentar. Se da certo acho que vai ser muito útil para meu trabalho, conheço sim as funções ditas, porém até agora não tinha conseguido rodar as análises usando múltiplas árvores, precisava saber um pouco de programação :)
Eu te escrevo assim que precisar alguma coisa.
2019/06/17 18:54